一个简明完整系统发育树构建流程!
1. 前言
构建一棵系统发育树是研究系统学和进化的基础。然而,发育树的构建是对分类单元进化历史的推测,因此对发育树的可靠性检验也是重中之重。一棵发育树的获得大致分为数据输入、数据处理 **、算法计算 、树的获取 、可靠性检验、树的可视化** 和树的注释等等。
数据输入一般分为两类,序列数据如DNA、AA、RNA,性状数据如形态数据、各种生态学数据。数据处理包括序列比对、饱和检验、碱基替换模型比较等。算法计算涉及到各种构建发育树的算法,如UPGMA、NJ、MP、ML、BI等,以及多基因数据的串联方法和溯祖理论之间的比较。树的获取指得是如何从多棵树中总结出一棵最优树。可靠性检验顾名思义,指对发育树拓扑结构的检验,包括自展支持、后验概率等,以及对不同发育树之间的比较。树的可视化是使用工具展示发育树以及使其更加美观的方法。树的注释包罗万象,如分化时间校准、祖先序列推测和各种生物地理学的各种注释,加深了树的深刻程度,和各种具体问题联系起来,使树的思想成为进化研究的基础方法。
Yang, Z. and Rannala, B., 2012. Molecular phylogenetics: principles and practice. Nature reviews. Genetics, 13(5), p.303.
2. 数据准备
2.1 序列比对
序列比对是系统发育树构建的基础,旨在找到理论上的同源位点。存在多种多样的比对算法,可以由不同软件来实现。常用的如Muscle、MAFFT等。此教程以Mega中的Muscle算法比对DNA非编码序列为例。
Wong, K.M., Suchard, M.A. and Huelsenbeck, J.P., 2008. Alignment uncertainty and genomic analysis. Science, 319(5862), pp.473-476.
-
打开Mega, 调出Align模块> edit/build Alignment > creat a new alignment > 选择DNA,出现Alignment explorer。所有的比对编辑操作均在这里面实现。界面如下:

-
通过edit导入新序列,edit> edit sequences form file

此处的file需要特定的格式,如常见的fasta(.fas)格式。格式如下:
>seq_1_name ATCT >seq_2_name ACCT >seq_3_name ACCG ......fasta格式比较简单,> 后跟随序列名称,不支持空格等大部分特殊字符,仅支持部分字符如_ . 等,序列另起一行。导入文件前,需要将测序得到的序列以此序列保存,扩展名 .txt, .fas均可。
-
序列导入以后即可比对,比对前界面如下:

然后 Muscle > Align DNA (如为编码序列,可截取完ORF以后,选择Align Codons),选择默认设置,点击compute即可。比对参数界面:

比对后则如下:

注:当序列明显无法比对整齐时,可检查是否为反向互补序列。
比对后的序列两段常常不整齐,此比对导致的gap(即-)会部分影响发育树结果,两段大部分截取删除后,少部分gap可用‘‘?’’填充整齐。

-
按需保存和导出比对结果。
比对结果的保存优先使用fasta格式,而一般发育树构建软件通常使用nexus格式作为输入文件。Mega支持这两种格式的输出。

2.2 碱基替换模型选择
碱基替换模型是对碱基突变的量化描述,一般为一个4*4的矩阵。
| A | G | C | T | |
|---|---|---|---|---|
| A | 0 | a | b | c |
| G | a | 0 | d | e |
| C | b | d | 0 | f |
| T | c | e | f | 0 |
4个碱基的突变情况,共产生6个不同的速率,这是一个最一般的模型,称为 General time reverse model,即GTR,该模型具有普适性,符合绝大部分数据集。该模型假设A – T和T – A具有相同的速率,即突变没有时间方向性,是可逆的。此等假设并未考虑其是否符合生物学的本质,仅仅是计算可行性上的需要。
该一般模型的种种简化,会产生各种模型,如F81、JC69、HKY等。一个重要的简化是把6个速率分为两类,即转换和颠换两种不同的速率也就是HKY模型。一般认为转换比颠换更为容易。
指的一提的是,一条序列上的全部位点并不具有完全相同的替换矩阵,即不同位置的碱基有着不同的突变速率,对于这种异质的突变速率通常通过Gamma分布(G)来描述,Gamma分布是一个灵活而强大的分布,具有较好的计算性。此外可能仍有部分碱基很少有突变事件发生,这些碱基会共同由一个不变位点比例(I)来描述。
(此段存疑)一般认为替换模型估算的突变数量仅和枝长相关,不影响拓扑结构。不同模型对拓扑结构的影响并不大,然而在计算时间节点之类对枝长敏感的算法时模型的选择和预设就更为重要。
2.2.1 PAUP + MrModelTest模型选择法
-
输入文件需要nexus (*.nex)格式,该格式可以由Mega比对完成后生成。首先用PAUP软件读取nexus格式文件。打开PAUP > 选择nexus文件。

-
运行模型计算批处理文件。PAUP中 File > open > mrblock.dat 运行自动进行,生成mrmodel.scores文件,该文件通常和mrblock.dat,modelGUI.exe一同位于MrMTGUI文件夹下。

Mrblock.dat会对24个模型分别计算其得分,得分记录于mrmodel.scores中。
-
最优模型评价。打开ModelGUI.exe > 点击右侧select file… > 选择mrmodel.scores文件 > 点击右侧MrModeltest!!!。界面如下:

不同模型间的比较评价遵循特定的标准,如hLRTs,AIC,AICc,BIC,BF等等。MrModeltest仅支持前两种评价标准,通常选用AIC标准。结果重点解读最优模型及其参数,自动生成的ML,BI的命令模块。
最优模型:
----------------------------------------------------- * * * AKAIKE INFORMATION CRITERION (AIC) * * * ----------------------------------------------------- Model selected: HKY+G -lnL = 1489.4283 K = 5 AIC = 2988.8567 Base frequencies: freqA = 0.1933 freqC = 0.3464 freqG = 0.3042 freqT = 0.1561 Substitution model: Ti/tv ratio = 2.5316 Among-site rate variation Proportion of invariable sites = 0 Gamma distribution shape parameter = 0.2838 包括各种参数如-lnL, AIC值,碱基频率,碱基替换速率,Gamma shape参数以及不变位点比例等。
贝叶斯命令模块:
BEGIN MRBAYES; Lset nst=2 rates=gamma; Prset statefreqpr=dirichlet(1,1,1,1); END; 如构建贝叶斯发育树,此段命令直接贴入nexus文件末尾即可。
ML命令模块(此处仅针对Garli软件):
BEGIN PAUP; Lset Base=(0.1933 0.3464 0.3042) Nst=2 TRatio=2.5316 Rates=gamma Shape=0.2838 Pinvar=0; END; 同理,此命令也需贴入nexus文件末尾。
2.2.2 jModeltest法选择模型
MrModeltest仅在24个模型中选择,Modeltest也不过是48个,这可能并不能选择到最优模型。Jmodeltest支持多达88个甚至1624个模型比较。jModeltest具有友好的图形界面,操作简单,但极耗硬件资源,对于较大的数据集几乎不能完成计算。简要操作如下:
-
打开jModeltest,File > Load DNA alignment, 输入数据,此处使用fasta格式。

-
计算模型得分。Analysis > compute likelihood scores > compute likelihood(选择默认参数,如有需要可自行修改),等待计算完成。


-
模型评价。jModeltest支持多种方法评价模型,一般来说BIC优于AIC,然而AIC更为常用。jModeltest 的结果可以保存到HTML格式使用浏览器查看。

结果可以在result > show results table 中查看,红色标记的即为最优模型。

2.2.3 形态数据的模型选择
除了较为常用的DNA序列的碱基突变模型选择,氨基酸数据的突变模型复杂的多,此处并不涉及。考虑到越来越多的形态数据的构树,形态数据的模型选择仍然需要考虑,然而以上两种方法均不支持。此处接好ModelFinder来评估其模型选择。
待续…
3. 系统发育树构建
系统发育树构建方法通常分为两类,基于距离的方法和基于性状的方法。
基于距离的方法是系统学早期发展起来的,将序列转化为距离矩阵然后根据距离矩阵构建聚类树,优点是速度极快,缺点很多,模型考虑简单,不适合远缘序列,不适合复杂序列,理论上不总是可以得到一个最优树。
基于性状的方法是系统发育的主流,不转化为距离矩阵,避免了数据的丢失,直接基于碱基序列计算。常见的包括最大简约法、贝叶斯法和最大似然法。
最大简约法不基于任何假设,不进行模型描述,认为具有最少突变步骤的发育树是最优树,计算强度较小,缺点同样是不适合远缘序列,无法考虑到复杂的突变事件。
最大似然法的基础是统计学的最大似然估计,把拓扑结构和枝长均视为参数,使观测数据(即碱基序列)有最大的似然值的参数为最优参数,即最优树。缺点是计算强度较大,可能会得到次优树。
贝叶斯方法则刚刚相反,基于观测数据,得分最高的拓扑结构被认为是最优树。蒙特卡洛(MC)和马尔科夫链(MC)的引入使得贝叶斯方法的得到极大的发展。贝叶斯方法具有较快的运算速度,多个链同时运行也可较大限度的避免局部最优化,因此被认为是最好的发育树构建方法。
3.1 最大简约法
最大简约法一般通过PAUP来实现,操作较为简单。
-
准备nexus文件,可通过Mega导出。nexus文件末尾需添加命令模块(block)。
begin paup; log file=****.log; set autoclose =yes; set criterion = parsimony; set root = outgroup; set storebrlens = yes; set increase=prompt; outgroup *** ***; bootstrap nreps=1000 search=heuristic conlevel=50 / addseq=random nreps=10 swap=tbr hold=1; savetrees from=1 to=1 file=****.tre format= altnex brlens = yes savebootp = NodeLabels MaxDecimals=0; pscores /TL=yes CI=yes RI=yes RC=Yes HI=Yes; end;此命令需要修改三处命令,1,指定log文件名,建议与nexus文件一致;2,指定外类群,多个外类群通过空格隔开;3, 指定tre文件名,同样建议和nexus文件一致。
-
PAUP直接打开保存好的nexus文件,就可直接运算。当bootstrap replicates达到预置的重复数时,程序结束。生成log文件和tre文件。

-
在此,我们涉及到两个概念,nexus文件和外类群,我们对它稍做解释。
nexus文件刚才选模型时已有所接触,此处再次解释。nexus和fasta文件一样是系统发育处理中常见的格式,但远比fasta复杂,可以记录序列文件和树文件。一个典型的nexus文件如下:
#NEXUS begin taxa; dimensions ntax=12; taxlabels Lemur_catta Homo_sapiens Pan Gorilla Pongo Hylobates Macaca_fuscata M._mulatta M._fascicularis M._sylvanus Saimiri_sciureus Tarsius_syrichta ; end; begin characters; dimensions nchar=898; format missing=? gap=- matchchar=. interleave=yes datatype=dna; options gapmode=missing; matrix Lemur_catta AAGCTTCATAGGAGCA Homo_sapiens AAGCTTCACCGGCGCA Pan AAGCTTCACCGGCGCA Gorilla AAGCTTCACCGGCGCA Pongo AAGCTTCACCGGCGCA Hylobates AAGCTTTACAGGTGCA Macaca_fuscata AAGCTTTTCCGGCGCA M._mulatta AAGCTTTTCTGGCGCA M._fascicularis AAGCTTCTCCGGCGCA M._sylvanus AAGCTTCTCCGGTGCA Saimiri_sciureus AAGCTTCACCGGCGCA Tarsius_syrichta AAGTTTCATTGGAGCC Lemur_catta CTGTCTAGCCAACTCT Homo_sapiens CTGCCTAGCAAACTCA Pan CTGCCTAGCAAACTCA Gorilla CTGCCTAGCAAACTCA Pongo CTGCCTAGCAAACTCA Hylobates CTGCCTTGCAAACTCA Macaca_fuscata CTGCCTAGCCAATTCA M._mulatta CTGCCTAGCCAATTCA M._fascicularis CTGCTTGGCCAATTCA M._sylvanus CTGCTTGGCCAACTCA Saimiri_sciureus CTGCCTAGCAAACTCA Tarsius_syrichta TTGCCTAGCAAATACA begin assumptions; charset coding = 2-457 660-896; charset noncoding = 1 458-659 897-898; charset 1stpos = 2-457\3 660-896\3; charset 2ndpos = 3-457\3 661-896\3; charset 3rdpos = 4-457\3 662-.\3; exset coding = noncoding; exset noncoding = coding; usertype 2_1 = 4 [weights transversions 2 times transitions] a c g t [a] . 2 1 2 [c] 2 . 2 1 [g] 1 2 . 2 [t] 2 1 2 . ; usertype 3_1 = 4 [weights transversions 3 times transitions] a c g t [a] . 3 1 3 [c] 3 . 3 1 [g] 1 3 . 3 [t] 3 1 3 . ; taxset hominoids = Homo_sapiens Pan Gorilla Pongo Hylobates; end; begin paup; constraints ch = ((Homo_sapiens,Pan)); constraints chg = ((Homo_sapiens,Pan,Gorilla)); end;通常nexus包括文件说明头:
#NEXUS begin taxa;矩阵维度说明:
dimensions ntax=12; taxlabels Lemur_catta Homo_sapiens ...... ; end; begin characters; dimensions nchar=898;字符串说明:(包括是否数据分段,此处为分段)
format missing=? gap=- matchchar=. interleave=yes datatype=dna; options gapmode=missing;序列矩阵:
Lemur_catta AAGCTTCATAGGAGCA Homo_sapiens AAGCTTCACCGGCGCA ...... Lemur_catta CTGTCTAGCCAACTCT Homo_sapiens CTGCCTAGCAAACTCA ......除了以上基本模块,还有一些其他的block块。例如数据分段(按基因、按密码子第几位等):
begin assumptions; charset coding = 2-457 660-896; charset noncoding = 1 458-659 897-898; charset 1stpos = 2-457\3 660-896\3; charset 2ndpos = 3-457\3 661-896\3; charset 3rdpos = 4-457\3 662-.\3; exset coding = noncoding; exset noncoding = coding;算法命令模块:(例如上文提到的PAUP的MP命令block,下面列出的替换模型模块以及单系约束命令,当然这些block并非所有的程序均支持。)
usertype 2_1 = 4 [weights transversions 2 times transitions] a c g t [a] . 2 1 2 [c] 2 . 2 1 [g] 1 2 . 2 [t] 2 1 2 . ; usertype 3_1 = 4 [weights transversions 3 times transitions] a c g t [a] . 3 1 3 [c] 3 . 3 1 [g] 1 3 . 3 [t] 3 1 3 . ; taxset hominoids = Homo_sapiens Pan Gorilla Pongo Hylobates; end; begin paup; constraints ch = ((Homo_sapiens,Pan)); constraints chg = ((Homo_sapiens,Pan,Gorilla)); end;此外nexus也可以保存树,如常见的.tre扩展名的文件实际上就是nexus的格式,例如:
#NEXUS Begin trees; [Treefile saved Thu Oct 05 11:34:24 2017] [! >Data file = C:\Users\zz\Desktop\8.nex >Bootstrap method with heuristic search: > Number of bootstrap replicates = 1000 > Starting seed = 1945405146 > Optimality criterion = parsimony > Character-status summary: > Of 646 total characters: > All characters are of type 'unord' > All characters have equal weight > 547 characters are constant > 69 variable characters are parsimony-uninformative > Number of parsimony-informative characters = 30 > Gaps are treated as "missing" > Starting tree(s) obtained via stepwise addition > Addition sequence: random > Number of replicates = 10 > Starting seed = 1945405146 > Number of trees held at each step during stepwise addition = 1 > Branch-swapping algorithm: tree-bisection-reconnection (TBR) > Steepest descent option not in effect > Initial 'MaxTrees' setting = 100 > Branches collapsed (creating polytomies) if maximum branch length is zero > 'MulTrees' option in effect > Topological constraints not enforced > Trees are unrooted > > 1000 bootstrap replicates completed > Time used = 0.75 sec > ] > tree PAUP_1 = [&U] (Ficus_maxima_ITS,((((2009198_Ficus_pumila_ITS_AF7,2010058_Ficus_sarmentosa_ITS_AH3)65,(2009274_Ficus_sarmentosa_ITS_AV9,GBOWS0029_Ficus_pubigera_ITS_AL1)62)89,2010066_Ficus_pumila_ITS_AH4)93,(2009415_Ficus_sagittata_ITS_AX5,2014059_Ficus_hederacea_ITS_AP7)99)67,2009411_Ficus_laevis_ITS_AX4)0; > End;
另外一个概念是外类群,指定外类群的目的是为了置根,置根有多种方法,如中点置根法、分子种赋根法,当然也还有一些较新的方法。置根之后的发育树才有方向,才可以看到祖裔关系。这里我们关注的外类群的挑选规则:外类群应该是所有内类群的姐妹群,关系越近越好。亲缘关系较远的外类群容易与内类群形成长枝吸引。
Tria, F.D.K., Landan, G. and Dagan, T., 2017. Phylogenetic rooting using minimal ancestor deviation. Nature Ecology & Evolution, 1, pp.s41559-017.
3.2 最大似然法
最大似然法最初是为了解决简约法的长枝吸引而引入的一种系统发育重建方法,其理论基础是发展非常成熟的最大似然估计方法。一开始,在PhyML和Garli等软件中实现最大似然法较为耗时,其后以RXaML、IQ-tree为代表的执行快速自展的算法,极大地提高了运行速度,基本上是最快的系统发育方法。以RXaML-master为例介绍最大似然树的构建:
-
运行RXaML-master。其没有用户界面,需要在cmd(命令提示符)运行。拷贝程序至C盘,打开执行文件(*.exe)所在文件夹.

-
拷贝比对好的fasta文件至该文件夹下。

-
打开cmd(命令提示符)程序。输入cd fasta文件所在路径,然后回车。cd此处意为change directory。

-
输入程序参数,回车后即开始运行,结果在同一文件夹下。此处先示例,然后再介绍常用参数。



-
结果会生成多个.tre文件,选择bipartion.tre即可在各种可视化工具中调整美化。
-
参数简介如下
raxmlHPC -s 9.fas -m GTRGAMMAI -N autoMR -x 23155001098 -p 23100551098 -T 4 -o Ficus_maxima_ITS -f a -n result
- rxamlHPC 程序头调用rxaml程序。
- -s 输入文件参数,支持phylip、fasta格式。
- -m 模型参数,不同数据源支持不同的模型,常见为DNA的GTR系列模型,如GTRGAMMA,GTRCAT,GTRGAMMAI等等。不支持非GTR模型。具体参见软件说明书。
- -N 自展重复参数,通常使用1000,也可以让软件决定多少为合适,参数为antoMR。
- -x, -p 随机数种子,可随意输入。
- -T 处理器线程数,仅在HPC版本及配置多线程环境后方才生效。
- -o 外类群参数,多个外类群间用, 隔开。
- -f 执行分析参数,a 此处为系统发育常用的快速自展分析。
- -n 输出结果扩展名参数,可任意指定。
3.3 贝叶斯方法
Mrbayes是实现贝叶斯算法的主要软件。
3.3.1 输入文件准备
Mega所导出的nexus的格式和贝叶斯所支持的nex格式略有区别。Mega所导出nexus前文已列出,下面列出Mrbayes的nex文件头:
begin data;
dimensions ntax=12 nchar=898;
format datatype=dna interleave=no gap=-;
matrix
差异主要包括三点:
-
修改begin taxa 为begin data
-
修改datatype=nucleotide至data=dna (仅针对DNA数据)
-
删除taxalabels模块
此外还包括文件末尾的Mrbayes模块。该模块灵活度很大,可简单也可复杂,最简单的情况仅为一个碱基突变模型指定模块(模型选择部分已提及),最复杂的情况可以包含从log文件、模型指定、运行参数指定、树总结参数指定等等。
一个相对详细的Mrbayes模块如下:
# Mrbayes block 模块,将此模块按照序列数据要求修改后,附于nex文件末尾即可。
# []内为每行参数的注释内容,可保留,软件默认不读取[]内的内容。
begin mrbayes;
[writting to log file]
log start filename=log.log;
[constraint some taxa as monophyly]
constraint outgroups = 36 37 157 158;
[designate outgroups as taxa index]
outgroup 36;
outgroup 37;
outgroup 157;
outgroup 158;
[designate data partitions as start and end points]
CHARSET its = 1-721;
CHARSET ets = 722-1218;
CHARSET g3pdh = 1219-1985;
partition gene=3: its,ets,g3pdh;
set partition=gene;
[designate model parameters as partitions]
lset applyto=(1) nst=6 rates=invgamma;
lset applyto=(2) nst=6 rates=gamma;
lset applyto=(3) nst=6 rates=invgamma;
Prset applyto=(all) statefreqpr=dirichlet(1,1,1,1);
[designate model parameter when data not partition]
[lset nst=6 rates=invgamma;
Prset statefreqpr=dirichlet(1,1,1,1);]
[designate mcmc chain parameters]
mcmcp ngen=15000000 samplefreq=1000 nruns=4 checkpoint=yes checkfreq = 10000 stoprule=yes stopval=0.01;
[execute mcmc]
mcmc;
[summary tree and postiror prabability after mcmc runs]
sumt relburnin=yes burninfrac=0.25;
sump relburnin=yes burninfrac=0.25;
end;
各命令解释:
-
log start filename:指定运行过程中的log文件名;
-
constraint outgroups:约束单系类群,后接类群序号,可指定多个单系群,另起一行继续写即可,但当想约束多个外类群为单系时,软件不生效;
-
outgroup:指定外类群,后接类群序号,可指定多个类群;
-
lset: 模型指定参数,包括若干子参数:
- nst:碱基突变速率个数,在1,2,6之间选择;
- 1时,对应JC、F81等模型;
- 2时,对应K80、HKY等模型;
- 6时,对应TrN、K81、GTR等模型;
- rates:突变速率模型等等;
- +G时,为gamma
- +I时,为pinv
- +I+G时,为invgamma
- nst:碱基突变速率个数,在1,2,6之间选择;
-
Prset:突变速率先验分布类型,Mrbayes中使用狄利克雷分布作为其先验;
-
mcmc:蒙特卡洛-马尔科夫链参数,包括若干重要子参数:
- ngen:链的代数;
- samplefreq:取样频率,太高则最终取样的树太少,不足以估算后续的树枝长和后验概率参数,太小则计算压力大且不容易收敛,一般1000-10000之间;
- nruns:同时独立运行的分析个数,默认为2,一般选择4;
- checkpoint:检查点,设置为yes时软件会定时记录当前运行状态,中断恢复运算时会从此状态恢复,Mrbayes 3.2之后此参数默认为yes
- checkfreq:检查频率,默认2000,可设置为10000;
- stoprule:终止规则,设置为yes时,收敛值小于设定值时会自动停止,尽管总代数可能还没有达到设置的总代数(ngen)
- stopval:设置的收敛值阈值,和stoprule=yes配合使用;
- 此时设置各参数后会立刻运行,如果想仅设置参数而不立刻运行,可使用mcmcp参数代替mcmc;
-
sumt:summary tree的缩写,总结树,包括若干子参数:
- burnin:老化值,mcmc链在收敛前存在一段变动较为明显的过程,这个数据对发育树枝长和后验概率的估算影响很大,需要舍去,一般为总数的1/10-1/4。此处针对的时抽样后树的个数,如总代数1000000,抽样频率1000,安装1/4的比例来去掉老化值的话,则应输入250。为了简便,直接指定老化比例,使用参数relburnin=0.25指定舍去前25%的代数,免去计算抽样的过程。
- contype:一致树的类型,halfcompat为50%多数一致树(默认),allcompat为严格一致二叉树;
- conformat:一致树的格式,Figtree格式为默认,包含多个参数,simple较为简单,仅包括枝长和后验概率,为大多数软件所支持。注意:如iTol等可视化软件仅支持simple模式。
-
sump:summary probability的缩写,估算后验概率,参数同sumt。
-
CHARSET:对数据进行分段,每段可以单独指定碱基突变模型,一个分段模块如下:
CHARSET its = 1-721; CHARSET ets = 722-1218; CHARSET g3pdh = 1219-1985; partition gene=3: its,ets,g3pdh; set partition=gene;配合数据分段的模型分段指定的模块如下:
lset applyto=(1) nst=6 rates=invgamma; lset applyto=(2) nst=6 rates=gamma; lset applyto=(3) nst=6 rates=invgamma; Prset applyto=(all) statefreqpr=dirichlet(1,1,1,1);
3.3.2 软件操作
将准备好的nexus文件放入到Mrbayes程序文件夹下即可准备运行。
Mrbayes的运行操作分为两种,一种打开Mrbayes后手动逐行输入命令行,如外类群,链长等参数,此时不需要准备复杂的Mrbayes block。另一个即为前面的提到的准备一个详尽的Mrbayes block,预先根据数据提前指定好其参数,此时重复分析时就不用每次重复手动输入命令行。此外,第二种方法还有一个优势,即支持中断续跑,如果在分析过程中,程序意外中断,仅需在mcmcp 命令中加入 append=yes,如下:
[designate mcmc chain parameters]
mcmcp ngen=15000000 samplefreq=1000 nruns=4 checkpoint=yes checkfreq = 10000 stoprule=yes stopval=0.01 append=yes;
Mrbayes免安装,没有图形化界面,所有操作均通过输入操作命令行进行,主界面如下:

如果选择在nexus文件中已经准备好了详尽的Mrbayes Block,此时就可以直接输入exe name.nex,回车后即可等待运行结束,在当前文件夹下可见.tre格式的树文件。运行过程界面如下:

3.3.3 Mrbayes的收敛问题
Mrbayes由于使用MCMC算法,与上述两个系统发育软件有所不同,会存在一个收敛问题。Mrbayes软件使用Average standard deviation of split frequencies来判断收敛,默认每5000代计算一次该参数,如果该参数小于0.01,软件运行到预设代数则会自动停止,如果没有就会提示是否需要继续增加代数!
但是由于数据集的差别,即使增加到很大代数后,该参数仍然大于0.01,此时有以下几种方法可以尝试解决:
-
继续提高代数,直至时间成本难以承受,或者代数增加到30000000-50000000代(约数,在该代数下,大部分数据集都应该完成收敛;
-
Average standard deviation of split frequencies只是一种判断收敛的方法,对于MCMC算法的输出结果,ESS( Effective Sample Size )也是一个参见的判读收敛的方式,该方式可以在Tracer中实现。当代数已经足够大的时候,使用Tracer软件打开.p文件即可,如果所有的ESS都大于200时,即可认为运算已经收敛;

-
提高抽样频率:可在1000-10000之间选择,提高抽样频率可以加快收敛速度。同时最好加长运算链长,以保证最终有足够多的树用来总结树的拓扑结构和后验概率;
-
如果在Tracer中检查仍然没有收敛的趋势,就需要对运行参数进行调节了。可以调节的参数如下:
- temp参数:热链的温度,也就是热链冷链交换的频率,默认为0.1,可以稍微调高一些;
- Ngammacat:默认为4,可改为16,提交Gamma分布的离散化估计精细度,会明显提高运算强度;
-
指定一颗起始树:一般使用UPGMA树作为起始树,一颗合适的起始树可以避免由不恰当起始树造成的局部最优和收敛缓慢。起始树的格式如下:
[begin trees; tree usertree=(A:0.01,B:0.01,((C:0.01,D:0.01):0.01,E:0.01):0.01); end;]
4. 发育树的可视化
发育树的可视化较为复杂,分为多个层面,从仅仅把发育树本身展示出来,到对发育树本身进行颜色标识强调等,再到加上各种各样的注释数据,如物种信息、分布地信息、形态特征以及基因结构多种多样。
对于发育树本身的展示和强调可以在Figtree、Mega等常用软件中进行。
4.1 Figtree
Figtree是一个图形化的发育树可视化软件,使用较为简单。界面如下,可自行研究:
4.2 iTol
iTol是一个发育树可视化的在线工具,可以方便对发育树进行注释,可实现的效果如下:
 |
 |
 |
|---|---|---|
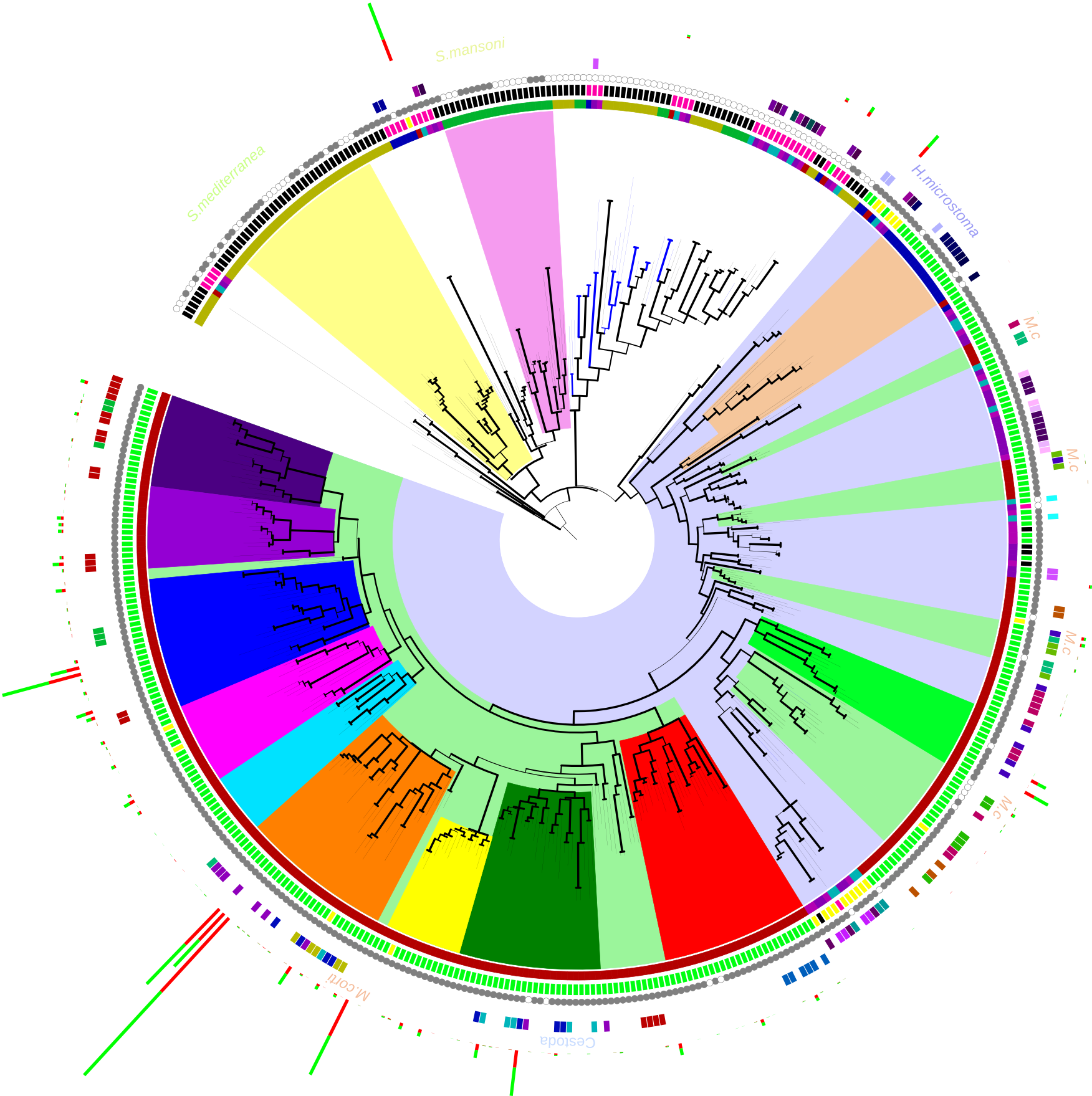 |
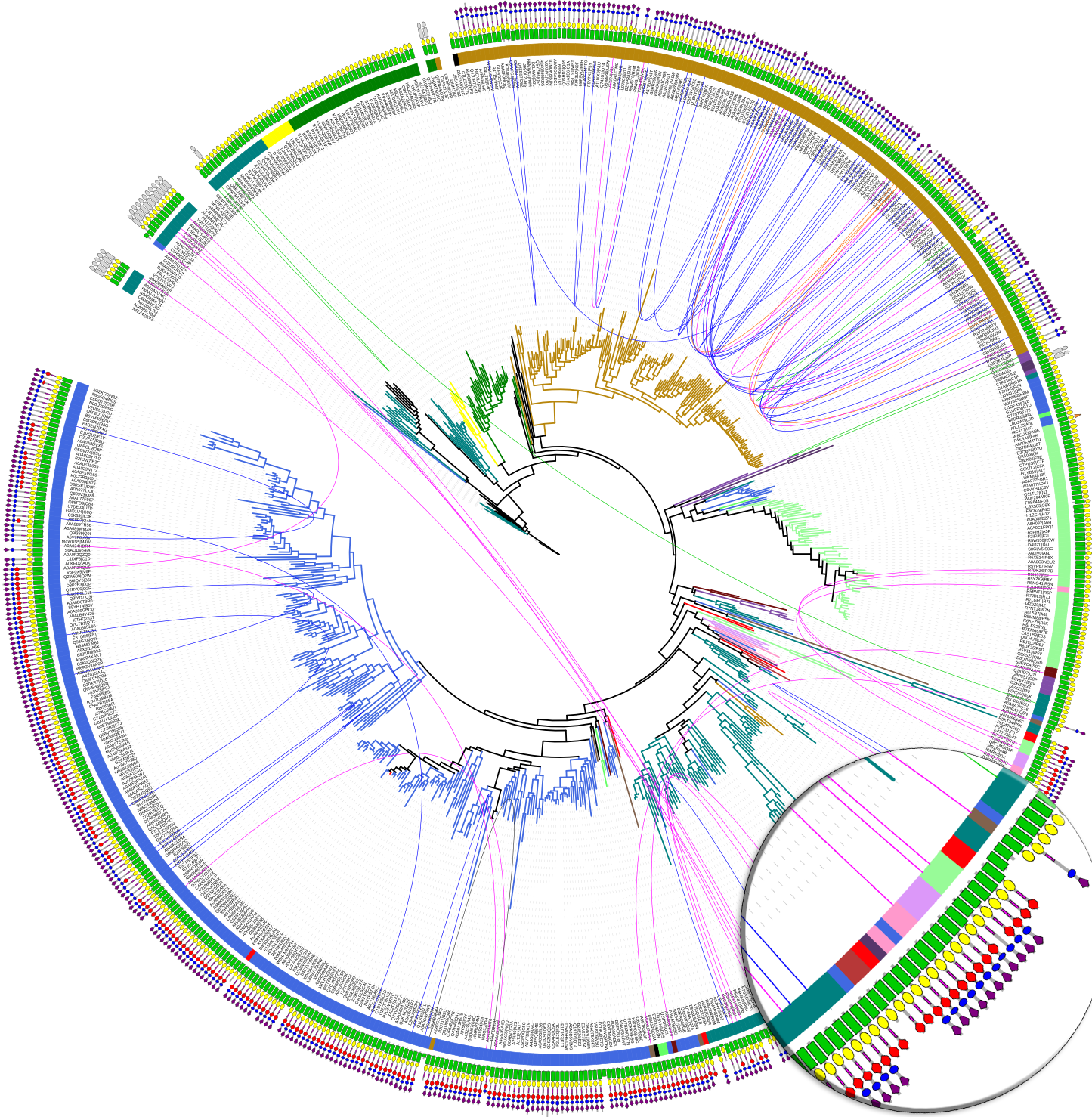 |
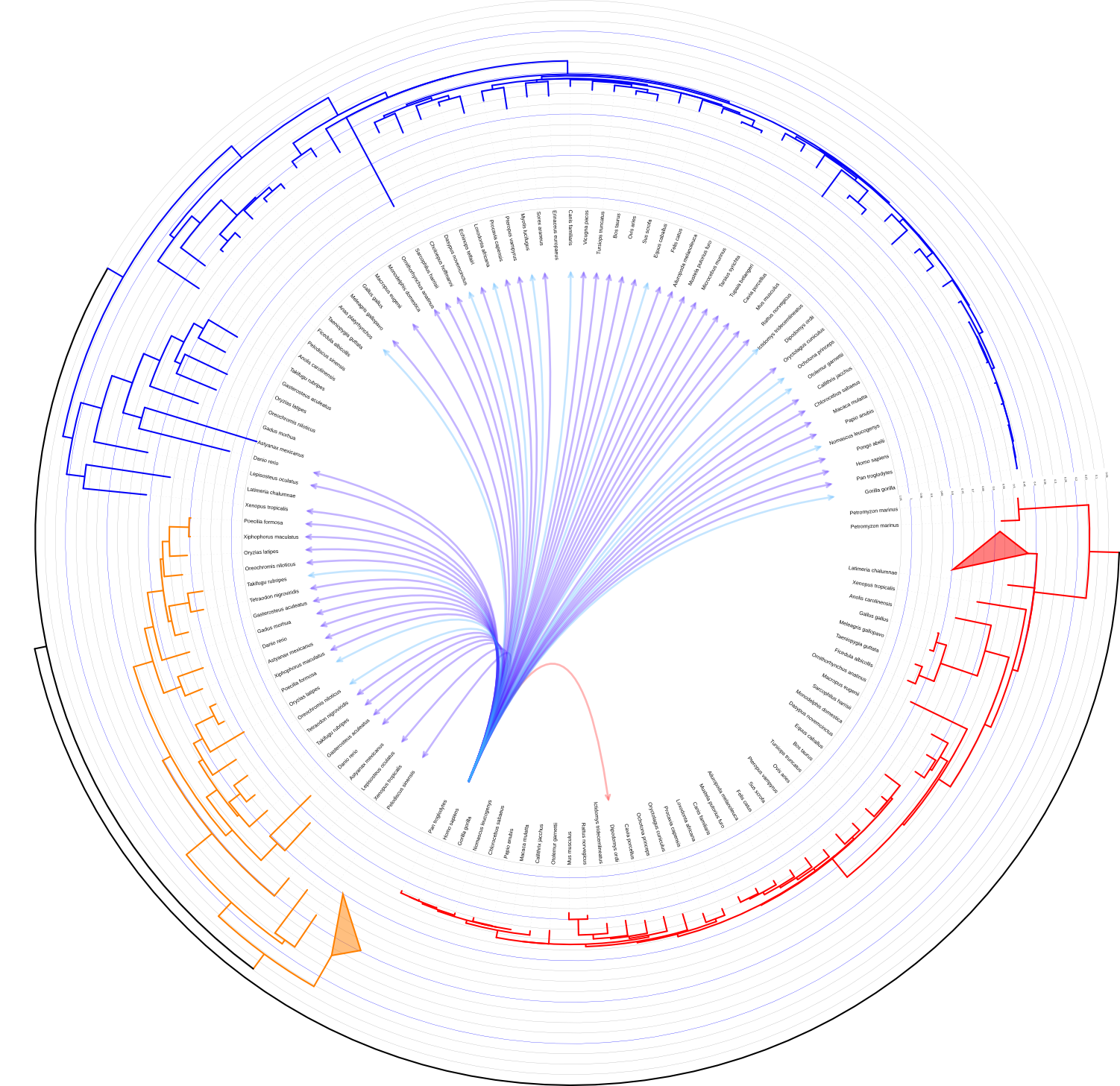 |
具体教程待续……
5. 发育树的检验
系统发育是一种历史过程,任何基于分子数据集得到的发育树都是对真实系统发生的推测。一个合理地假设,总是应该接收来自各种证据的检验。
系统发育假设检验(phylogenetic hypothesis testing)是用统计学方法检验两个或多个不同发育树的差异是否有统计学上的显著性。系统发育检验需要数据集、模型、两棵以上的发育树。已有有大量的检验方法,主要包括频率检验或者贝叶斯检验。一般来说,检验方法包括Approximately unbiased test,Approximate Bayesian posterior probability test,bootstrap probability test,Kishino-Hasegawa test,weighted Kishino-Hasegawa test,Shimodaira-Hasegawa test和weighted Shimodaira-Hasegawa test等。常用的为Approximately unbiased test (AU)和Kishino-Hasegawa test (KH)。
黄原. (2012). 分子系统发生学. 科学出版社. pp 381-393.
多个软件都可以用于执行这种检验,如 PAUP,TREE-PUZZLE等。此处,我们介绍consel 01j.
Shimodaira, H., & Hasegawa, M. (2001). CONSEL: for assessing the confidence of phylogenetic tree selection. Bioinformatics, 17(12), 1246-1247.
具体教程请点击这里!